BioSequence[type,"seq"]
配列"seq"に対応する指定の type の生体分子配列を表す.
BioSequence["seq"]
配列から(DNA,タンパク質等の)タイプを推測する.
BioSequence[ent]
遺伝子実体またはタンパク質実体の ent に関連付けられた生体分子配列を与える.
BioSequence[type,{chem1,chem2,…}]
type が与えられた化学物質のリストに対応する生体分子配列を与える.
BioSequence[type,"seq",{bond1,bond2,…}]
与えられた結合のリストを持つ生体分子配列を表す.
BioSequence["HybridStrand",{bioseq1,bioseq2,…},{bond1,bond2,…}]
共有のプライマリリンケージを持つ複数のモチーフ配列から構成された配列を表す.
BioSequence[{bioseq1,bioseq2,…},{bond1,bond2,…}]
追加的な結合のみで繋がれた配列の数を表す.


BioSequence
BioSequence[type,"seq"]
配列"seq"に対応する指定の type の生体分子配列を表す.
BioSequence["seq"]
配列から(DNA,タンパク質等の)タイプを推測する.
BioSequence[ent]
遺伝子実体またはタンパク質実体の ent に関連付けられた生体分子配列を与える.
BioSequence[type,{chem1,chem2,…}]
type が与えられた化学物質のリストに対応する生体分子配列を与える.
BioSequence[type,"seq",{bond1,bond2,…}]
与えられた結合のリストを持つ生体分子配列を表す.
BioSequence["HybridStrand",{bioseq1,bioseq2,…},{bond1,bond2,…}]
共有のプライマリリンケージを持つ複数のモチーフ配列から構成された配列を表す.
BioSequence[{bioseq1,bioseq2,…},{bond1,bond2,…}]
追加的な結合のみで繋がれた配列の数を表す.
詳細とオプション











- BioSequence[…]を評価すると,可能であれば以下の形式になる.
-
BioSequence[type,"seq",bonds] モチーフ(単一のタイプの単一の鎖) BioSequence["HybridStrand",{bioseq1,bioseq2,…},bonds] ハイブリッド鎖(複数のタイプの単一の鎖) BioSequence[{bioseq1,bioseq2,…},bonds] 配列コレクション(追加的な結合を持つたくさんの鎖) - BioSequenceは次の文字を使って各 type の分子を表す.
-
"DNA" A, C, G, T "CircularDNA" A, C, G, T "RNA" A, C, G, U "CircularRNA" A, C, G, U "Peptide" A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y "CircularPeptide" A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y - 以下の表のコンテンツは,例えばEntity["BioSequenceType","DNA"]["Alphabet"]のように,"BioSequenceType"実体の"Alphabet"特性を通して得ることができる,
- 次は,各DNA(RNA)文字に対応するヌクレオチドである.
-
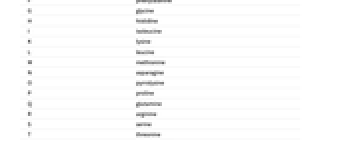
A アデニン C シトシン G グアニン T (U) チミン(ウラシル) - 同様に,以下は各ペプチド文字に対応するアミノ酸である.
-
A アラニン C システイン D アスパラギン酸 E グルタミン酸 F フェニルアラニン G グリシン H ヒスチジン I イソロイシン K リジン L ロイシン M メチオニン N アスパラギン O ピロリシン P プロリン Q グルタミン R アルギニン S セリン T トレオニン U セレノシステイン V バリン W トリプトファン Y チロシン - 上記の表のコンテンツは,例えばEntity["BioSequenceType","DNA"]["AlphabetRules"]のように,"BioSequenceType"実体の"AlphabetRules"特性を通して得ることができる.
- "Peptide"タイプと"CircularPeptide"タイプは,ピリオド(.)またはアスタリスク(*)を使って生体分子翻訳が停止した箇所を示すことができる.
- さらに,type をNoneにして与えられた化学的な意味がない遺伝子配列を表すことができる.
- BioSequenceは潜在的な化学物質の数を表す縮重記号を許容する.
- 次は,DNAおよびRNAに認められる縮重記号である.
-
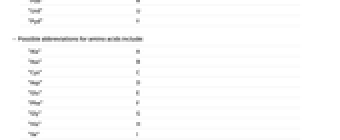
B C, G または T/U(Aではない) D A, G または T/U(Cではない) H A, C または T/U(Gではない) K G または T/U(ケトン) M A または C(アミノ) N A, C, G または T/U(任意の文字) R A または G(プリン) S C または G(強) V A, C または G (Tではない) W A または T/U(弱) Y C または T/U(ピリミジン) - 次は,ペプチドに認められる縮重記号である.
-
B D または N J I または L X A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y Z E または Q - 上記の表のコンテンツは,例えばEntity["BioSequenceType","DNA"]["DegenerateLetterRules"]のように,"BioSequenceType"実体の"DegenerateLetterRules"特性を通して得ることができる,
- タイプと長さが与えられた場合は,次の文字が任意の文字として使われる.
-
"DNA" または "CircularDNA" N "RNA" または "CircularRNA" N "Peptide"または "CircularPeptide" X - BioSequenceは,配列文字の部分に標準的な略字を使うことができる.
- 次はDNA塩基の使用可能な略字である.
-
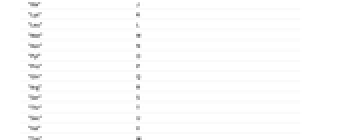
"dAdo" A "dCyd" C "dGuo" G "dNuc" N "dPuo" R "dThd" T "dPyd" Y - 次はRNA塩基の使用可能な略字である.
-
"Ado" A "Cyd" C "Guo" G "Nuc" N "Puo" R "Urd" U "Pyd" Y - 次はアミノ酸の使用可能な略字である.
-
"Ala" A "Asx" B "Cys" C "Asp" D "Glu" E "Phe" F "Gly" G "His" H "Ile" I "Xle" J "Lys" K "Leu" L "Met" M "Asn" N "Pyl" O "Pro" P "Gln" Q "Arg" R "Ser" S "Thr" T "Sec" U "Val" V "Trp" W "Xaa" X "Tyr" Y "Glx" Z - 配列によって暗示される接続に加え,BioSequence文字は追加的なBond実体でも接続できる.
- Bond[{i,j},type]の形で指定された結合は,文字列中の位置 i と j に対応する化学物質をタイプ type の結合で接続する.例えば,DNA配列"ACCT"の"A"と"T"を接続する水素結合はBioSequence["DNA","ACCT",{Bond[{1,4},"MultiHydrogen"]}]と表すことができる.
- 配列レベルの単一の結合で分子レベルの複数の結合を表すことができる.前の例では,"A"と"T"の間のBondが分子レベルの2つの水素結合を表している.
- ハイブリッド鎖では,Bond[{{i1,i2},{j1,j2}},type]の形の結合が,それぞれ位置 i2と位置 j2にインデックス i1とインデックス j1を持つモチーフ鎖を type で指定された結合で繋ぐ.例えば,DNA/RNAのハイブリッド配列 {"ACC","CCU"}の"A"と"U"を繋ぐ水素結合はBioSequence["HybridStrand",{"ACC","CCU"},{Bond[{{1,2},{2,3}},"MultiHydrogen"]}]と表すことができる,
- 配列コレクションでは,Bond[{{i1,i2,i3},{j1,j2, j3}},type]の形の結合が,位置 i3と位置 j3のインデックス{i1,i2}とインデックス{j1,j2}を持つモチーフ鎖をタイプ type の結合を介してそれぞれ結合している.
- モチーフ鎖が配列コレクションレベルで結合されようとしているなら,{i1,1,i3}または{i1,i3}が使われる.例えば,"CAC"と"CTC"という2つのDNA配列が与えられると,最初の配列の"A"と2番目の配列の"T"を繋ぐ水素結合は BioSequence[{"CAC","CTC"},Bond[{{1,1,2},{2,1,2}},"MultiHydrogen"]]またはBioSequence[{"CAC","CTC"},Bond[{{1,2},{2,2}},"MultiHydrogen"]]と表すことができる.
- 配列コレクションの中のハイブリッド鎖については,すべてのインデックスが必要である.例えば,DNA/RNAのハイブリッド配列{"ACC","CCU"}が配列コレクションの4番目の配列だとすると,"U"を参照する結合インデックスは{4,2,3}になる.
- DNAとRNAの配列文字はすべて"MultiHydrogen"結合タイプで繋ぐことができる.
- ペピチド配列では,すべての結合タイプがすべての配列の化学物質に適用される訳ではない.次の結合タイプは表示されたペプチド文字だけを結合する.
-
"DisulfideBridges" C ↔ C, U ↔ U, C ↔ U "LactamBridges" D ↔ K, E ↔ K - 例えば,BioSequence["Peptide","CGGGU",Bond[{1,5},type]]の type は"DisulfideBridges"でもよいが"LactamBridges"ではならない.
- モチーフ鎖についての結合もまたドット・ブラケット記法で入力できる.この形式は配列の結合を文字列の各文字が文字列中の位置に対応する単一の文字列として表す.結合文字列の有効な文字は結合なしまたはカッコ((と ))を表すピリオド(.)またはネストした結合ペアを表す山カッコ(<と>)である.例えば,文字列"<((..>))."は9文字からなる配列に適しており,{Bond[{1,6}],Bond[{2,8}],Bond[{3,7}]}がそれに相当するだろう.
- BioSequence[…]["prop"]によって得られるBioSequenceの特性"prop"には以下がある.
-
"SequenceType" "BioSequenceType"実体としての配列タイプ "SequenceString" 配列を表す文字列 "SequenceBondList" 配列中の明示的に与えられたすべての結合のリスト "SequenceBondCount" 配列中の明示的に与えられた結合の数 "SequenceLength" 配列の長さ "SequencePattern" 縮重記号を展開した文字列式 "AbbreviationSequence" 使用可能な略字を使った配列表現 "ChemicalList" 文字通りの化学実体のリスト "ChemicalPatternList" 縮重記号に許される化学実体のリスト "MolecularMass" 配列の分子量 "MolarMass" 配列のモル質量 "HELM" 配列のHELM文字列 "Properties" 特性のリスト - "ChemicalList"と"ChemicalPatternList"はどちらも,配列の各項に対して特定の化学物質を与える.前者は縮重記号をサポートしないが,後者はAlternativesを使って縮重記号を表す.
- 配列に縮重項がある場合は,その分子量がIntervalになる可能性がある.
- "HELM"特性はBioSequenceの巨大分子表現の階層的編集言語を与える.
- BioSequenceの可能なタイプは,"ExtendedBioSequenceType"実体でEntityStoreを作り,それを登録(EntityRegister)して拡張することもできる.
- 次は,定義可能な"ExtendedBioSequenceType"特性である.
-
"Alphabet" この配列内で認められている文字のリスト "AlphabetRules" 文字から特定の化学物質への連想 "BibliographicSource" 配列タイプについて記述している外部識別子 "Caption" フォーマットされた出力で配列の上に付くキャプション "ComplementLetterRules" 補体操作を定義する双方向規則 "Icon" 配列のフォーマットされた出力に表示されるアイコン "MolecularMassRules" 文字から分子量への連想 - "Icon"は,画像または既存の配列タイプの正規名として与えられる.
- "MolecularMassRules"は"AlphabetRules"を介して与えられる化学物質の分子量を無効にし,化学物質が与えられていない場合の分子量の計算を可能にする.
- BioSequenceQ[bioseq]は,bioseq が有効なBioSequence式に対応するときにのみTrueを与える.
例題
すべて開く すべて閉じるスコープ (28)
基本的な配列 (8)
実体からの配列 (4)
生体分子配列を構築するときは,"BioSequenceType"実体をタイプとして使うことができる:
結合のある配列 (4)
Bondを使って配列に追加的な構造を加えることができる:
ハイブリッド鎖 (5)
特性と関係 (28)
BioSequenceは特性数を与える:
BioSequenceのタイプは配列を説明するさらに多くの特性を含む実体である:
可能な縮重選択によって変化するオリゴヌクレオチド(つまり,一本鎖)分子量にアクセスする:
ハイブリッド鎖のほとんどの特性はもとになっているモチーフ配列の特性リストである:
配列コレクションの特性のほとんどはもとになっているモチーフ配列のリストのリストである:
"MolecularMass"特性と"MolarMass"特性はハイブリッド鎖全体に適用される:
与えられたタイプの基本記号は,"BioSequenceType"実体の"Alphabet"特性に対応する:
BioSequenceモチーフはMoleculeへの入力として与えることができる:
ハイブリッド鎖のBioSequenceはMoleculeへの入力としても与えることができる:
BioSequenceのコレクションもまたMoleculeに与えることができる:
ConnectedMoleculeComponentsを使って配列コレクションの別々の分子を得ることができる:
SequenceAlignmentは,2つのBioSequenceの例の間のアライメントを求めることができる:
RandomInstanceは,縮重したBioSequenceから完全に指定された例のサンプルを取ることができる:
BioSequenceQは,BioSequenceが指定タイプである,あるいはこれが他の属性を持つことを検証できる:
BioSequenceComplementおよびBioSequenceReverseComplementはBioSequenceの遺伝的補体を求める:
BioSequencePlotはBioSequenceの図表を与えることができる:
タイプが"DNA","RNA","CircularDNA"あるいは"CircularRNA"のBioSequenceをMoleculeに変換する際,配列は5' 3'の方向(プラス・センス)に進むものと解釈される:
タイプが"Peptide"あるいは"CircularPeptide"のBioSequenceをMoleculeに変換する際,配列はN末端からC末端に進むものと解釈される:
考えられる問題 (4)
指定されたタイプについて定義できない記号を含む配列はフォーマットされない:
指定の文字列に適した配列タイプを推測することはできないかもしれない:
すべてのハイブリッド鎖がMoleculeに変換できる訳ではない:
ハイブリッド鎖に非互換のモチーフタイプがある場合も質量特性が解釈できない原因となる:
標準的な略語がすべてのDNAおよびRNAの文字について定義されているわけではない:
おもしろい例題 (3)
ヒトのインスリンをBioSequenceとして表す:
Moleculeに変換する:
関連項目
BioSequenceQ BioSequenceComplement BioSequenceReverseComplement BioSequenceTranslate BioSequenceBackTranslateList BioSequenceTranscribe BioSequenceModify BioSequenceInstances BioSequencePlot
実体タイプ: BioSequenceType
Function Repository: BioSequenceGraph BioSequenceToRegularExpression BioSequenceMoleculePlot BioSequenceFoldingMatrix GetPubMedIdentifiers PeptideName NucleotideName NucleotideStaveDiagram NuIDEncode NuIDDecode RemoveDegenerateSequenceDifferences RNAFoldingMaximumBasePairing
関連するガイド
-
▪
- 生命科学と医学のデータと計算 ▪
- 配列アラインメントと比較 ▪
- 生体分子配列
テキスト
Wolfram Research (2020), BioSequence, Wolfram言語関数, https://reference.wolfram.com/language/ref/BioSequence.html (2022年に更新).
CMS
Wolfram Language. 2020. "BioSequence." Wolfram Language & System Documentation Center. Wolfram Research. Last Modified 2022. https://reference.wolfram.com/language/ref/BioSequence.html.
APA
Wolfram Language. (2020). BioSequence. Wolfram Language & System Documentation Center. Retrieved from https://reference.wolfram.com/language/ref/BioSequence.html
BibTeX
@misc{reference.wolfram_2025_biosequence, author="Wolfram Research", title="{BioSequence}", year="2022", howpublished="\url{https://reference.wolfram.com/language/ref/BioSequence.html}", note=[Accessed: 03-March-2026]}
BibLaTeX
@online{reference.wolfram_2025_biosequence, organization={Wolfram Research}, title={BioSequence}, year={2022}, url={https://reference.wolfram.com/language/ref/BioSequence.html}, note=[Accessed: 03-March-2026]}